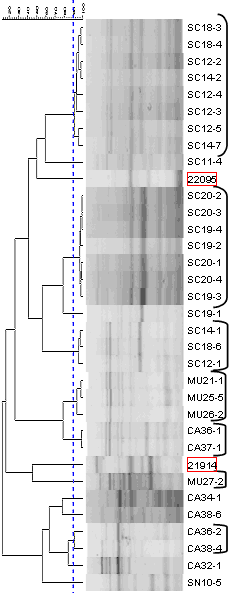
 |
| ERIC-PCR de cepas nasales de Haemophilus parasuis obtenidas de 3 granjas diferentes. Los aislados bacterianos con el mismo patrón de bandas son una misma cepa (señalados con flechas del mismo color). |
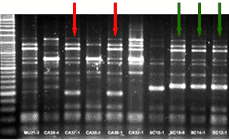 |
| Los patrones de ERIC-PCR se pueden analizar con programas especiales que permiten comparar los diferentes aislados. Diversas cepas nasales se compararon entre ellas y con cepas aisladas de lesiones (en recuadros rojos). Los patrones se consideran iguales hasta un nivel del 90% de identidad (línea punteada azul). |
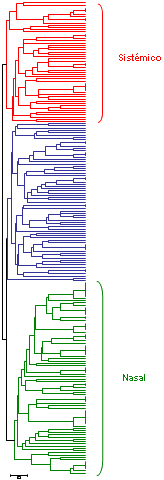 |
| Clasificación de 211 cepas de Haemophilus parasuis mediante MLST. Se observa un grupo asociado a cepas aisladas de lesiones sistémicas (en rojo), otro grupo asociado con cepas aisladas de nariz de animales sanos (en verde) y un último grupo intermedio (en azul). |
El cerdo es el único hábitat natural de Haemophilus parasuis, que además carece de capacidad para sobrevivir en el medio ambiente. Esto hace que la transmisión de esta bacteria sea siempre a través del contacto con animales colonizados o infectados por ella. Así, los lechones reciben la bacteria de sus madres y H. parasuis puede aislarse como colonizador del tracto respiratorio superior de animales sanos a los pocos días de vida. En consecuencia, la prevalencia de H. parasuis en granjas convencionales es prácticamente del 100%, aunque la incidencia de la enfermedad es mucho menor. En una granja convencional sana es normal aislar varias cepas de H. parasuis, que se mantienen en equilibrio con la inmunidad de los animales. Sin embargo, desequilibrios, como la introducción de nuevas cepas por mezcla de animales, pueden causar la aparición de enfermedad. Datos de EEUU indican una incidencia del 7% de la enfermedad de Glässer en el año 2000, que ha ido en aumento hasta un 17% en el año 2006.
Por lo tanto, es importante poder identificar las distintas cepas circulantes y diferenciar cepas meramente colonizadoras de aquellas capaces de producir enfermedad. Con este fin se han desarrollado varios métodos de clasificación de cepas o “tipado” de H. parasuis.
Los métodos de tipado pueden ser útiles en estudios epidemiológicos locales o globales. Los estudios locales se centran en analizar las cepas presentes en una granja o granjas relacionadas, determinar la introducción de nuevas cepas o comprobar si un tratamiento ha eliminado la cepa causante de un brote. Por su lado, los estudios globales analizan las relaciones de unas cepas concretas con otras aisladas en años y lugares distintos.
Clásicamente, las cepas de H. parasuis se han clasificado mediante serotipación. En 1992, Kielstein y Rapp-Gabrielson establecieron 15 serotipos en H. parasuis y determinaron la virulencia de las cepas de referencia de los distintos serotipos mediante infección experimental. Según esa clasificación, los serotipos más virulentos son el 1, 5, 10, 12, 13 y 14. En estudios realizados en distintos países, incluida España, se ha observado que los serotipos más prevalentes son el 5 y el 4; siendo el 4 de virulencia moderada. Aunque la serotipación tiene la ventaja de proporcionar información sobre la reacción antigénica de las cepas, su mayor problema es que existen una gran cantidad de cepas que no son tipables (entre 15 y 41%).
Para evitar este problema se comenzaron a desarrollar métodos de genotipado, que analizan material genético presente en todas las cepas. El mayor inconveniente del genotipado es la falta de información directa, por lo que las clasificaciones obtenidas tienen que complementarse con otros datos, como el origen clínico de la cepa. Existen varios métodos de genotipado, que se basan tanto en la comparación de perfiles de bandas como en la comparación de secuencias.
Un ejemplo de método de comparación de bandas utilizado en la clasificación de cepas de H. parasuis es la ERIC-PCR (“enterobacterial repetitive intergenic consensus” PCR). Esta técnica es útil en estudios locales de cepas de H. parasuis, pero su reproducibilidad no es buena y los resultados de distintos laboratorios no pueden compararse. Para mejorar el genotipado, se han desarrollado técnicas de secuenciación, como el “multilocus sequence typing” o MLST. Mediante MLST, las cepas de H. parasuis se clasificaron en varios grupos que presentaban relación con la virulencia potencial de las cepas. Un grupo incluía cepas asociadas a enfermedad sistémica y cepas virulentas de referencia, mientras que otro grupo incluía cepas nasales de animales sanos y cepas de referencia no virulentas.
Los métodos de genotipado tienen un gran potencial para la realización de estudios epidemiológicos de H. parasuis, pero se requieren más esfuerzos para relacionar la clasificación genómica y la virulencia real de las cepas.




